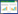2. 系统生物工程教育部重点实验室, 天津 300350;
3. 天津大学化工协同创新中心合成生物学平台, 天津 300350
2. Key Laboratory of Systems Bioengineering of Ministry of Education, Syn Bio Research Platform, Tianjin University, Tianjin 300350, China;
3. Collaborative Innovation Center of Chemical Science and Engineering, Tianjin University, Tianjin 300350, China
随着大数据和人工智能的不断发展,人们逐步认识到数据和算法将成为企业乃至国家生产力、竞争力的核心之一。然而,数字信息的产生和储存方式正在发生着范式转变,全球产生的数据量正呈指数式增长[1]。磁性(如磁带或硬盘驱动器)、光学(如蓝光)和固态(如闪存)等现有的主流存储设备将难以满足大规模数据的存储需求。而DNA作为一种与现有任何存储介质完全不同的数字材料,由于存储密度高(1018 byte·mm-3)[2-4]、存储时间长(数千年)[5, 6]、能耗低等优点,近年来被广泛关注,在海量冷数据存储方面,有望成为一种低成本的、稳定的存储解决方案。
经过十几年的发展,DNA数据存储的核心概念,包括高通量DNA合成[7-12]、碱基编解码算法[13, 14]、无错数字信息解码[15]和长期稳定保存[16]等都得到了验证以及形成了一定的技术积累。然而,现有报道的DNA存储技术系统所展示的数据存储能力与实际数据存储系统之间都存在着巨大的鸿沟。首先,虽然DNA的理论信息密度非常高,但DNA数据存储系统的物理存储密度实际上受到了保存介质的限制。现有技术大部分是基于已有的分子生物实验体系构建并验证的,而为分子生物技术发展起来的生化反应系统很难实现整体规模的放大,例如,现有的生化实验用的试管等物理方法其操作很难支撑大规模的操作,大大限制了DNA的物理存储密度[17]。按照IDC的预测,全球的数据全部用1 TB的硬盘存储,需要超过1 750亿个,如果全部要光盘存储,所有光盘叠加起来的高度可绕地球444圈。以百度阳泉云计算中心为例,目前已上线的服务器超过15万台,占地面积12万m2,存储容量超过6 EB,若以目前DNA信息存储中最常用的液体法——试管,以最大存储容量200 MB进行低温保存同样的数据,占用的空间以及维护的成本无法想象。其次,在访问数据的时候,需要对特定的数据进行检索和读取,而不需要对整个数据池进行访问,因此检索方式对DNA存储系统的实用性至关重要[14, 18],而DNA的保存形式也对存储于DNA中的文件的检索方式有至关重要的影响。最后,尽管当代方法大多采用自动化的合成[19, 20]和测序技术(例如,柱合成、阵列合成、Illumina、纳米孔等)[13, 14, 21, 22],但重要的中间步骤仍然主要是手动,DNA数据的写入与读取过程中仍存在大量繁琐操作,无法实现写、存、读整个流程的自动化,使其目前难以应用于大规模档案式数据的存储。总之,在面对海量数据存储时,DNA的保存、检索方式与工业应用的兼容性和实用性、集成的可操作性、数据的稳健性与安全性还存在很多问题。在保证较高的DNA物理存储密度的同时,可以实现设备的可管理化、对文件方便快捷的进行检索和访问。
本论文围绕DNA存储技术实现规模化和实用化需要的文件检索和高度集成等技术,介绍了DNA承载介质对于构建集成化设备的影响,综述了目前适用于DNA数据存储的检索方式,并且对DNA信息存储设备集成的发展及可能性进行了论述,希望为适用于大规模数据的集成化、自动化设备的开发提供参考。
1 DNA承载介质氧化剂、烷化剂、紫外线以及高能电磁辐射如X射线等容易对DNA序列造成损伤,从而造成存储于其中的数字信息的丢失[23, 24]。目前已经有液状、干粉状、包封法以及细胞体内等多种保存方式,可大大延长DNA的保存时间。
DNA水溶液是很常见的DNA保存形式,但核酸对脱嘌呤[25]、脱嘧啶[26]、脱氨基[27]和水裂解[28]是非常敏感的,这限制了在水介质中长期稳定性的可能[29, 30]。DNA水溶液通常放置在EP管等容器中进行保存,当利用EP管进行单个文件的分隔时,容器的体积大小将大大限制DNA的物理存储密度,并且冰箱等低温设备的体积也进一步限制了物理存储密度[图 1(a)]。将DNA以干粉的形式进行保存也是常用的方法之一[28, 31],目前常以喷雾干燥、喷雾冷冻干燥、空气干燥或冷冻干燥等方式对于DNA进行脱水[32],但是DNA粉末同样需要放置在容器中进行存储,读取时将粉末溶解通过PCR扩增进行读取。因此,DNA干粉保存同样面临容器体积影响DNA物理存储密度的问题以及文件选择性读取的问题[图 1(b)]。
|
| 图 1 DNA保存方式 Fig.1 The preservation of DNA |
| |
包封法是指通过阻隔性能较强的材料将DNA进行包埋,使DNA与外界环境中的水分和氧气隔绝,实现DNA的长期稳定保存。苏黎世联邦理工学院的Grass团队使用二氧化硅[15]或MgCl2等碱金属盐[33]对DNA进行封装可将DNA保存时间延长至2 000年[图 1(c)],且将DNA载量提高至20%(质量分数,下同)以上。由于包封粒子的直径较小(25~200 nm),同样需要容器对粒子进行保存。并且DNA的载量仍然较低,影响了DNA的物理存储密度,需要开发更合适的材料,提高DNA的载量。
目前,已经实现将存储有数字信息的DNA以基因组或质粒的形式保存在细胞体内[图 1(d)][34-37]。相比于体外存储,细胞体内DNA存储的复制保真度更高、复制成本更低、保存时间更长,但是体内数字信息的存储总量偏低。Chen等[38]将254 KB的数据写入1条人工染色体中,并在酵母细胞体内实现了稳定复制。2020年,Qi等[39]利用混菌培养,成功将445 KB的数字信息存储于大肠杆菌细胞内,这是目前数据量最大的体内存储。相比于200 MB[14]的体外存储,445 KB的存储总量仍然很低,所以存储总量目前是限制体内物理存储密度的主要因素。与DNA溶液相同,细胞同样需要放置在EP管等容器中,并将容器置于冰箱等冷冻设备中保存。因此,体内存储同样面临容器及冷冻设备占用物理空间和维护冷冻设备成本较高的问题。
2 DNA数字信息检索方式在对DNA中存储的数据进行读取和解码时,通常需要对整个寡核苷酸池进行扩增和解码。但是面对海量信息存储时,这种检索方式显然是不切实际的,这大大浪费了测序及计算资源。为了实现低成本、更便捷的数据读取,我们希望能够仅检索特定的数据而无需对整个寡核苷酸池进行访问(表 1)。
| 检索方法 | 文件名称 | 文件大小/kB | 物理密度/(byte·mm-3) | 检索方式 |
| Boolean search | 20个图像文件 | 2 | 6.6×1015 | 唯一引物ID |
| QR code | Hunminjeongum文件 | 135 | 1.0×1012 | 元数据访问 |
| DMF and silica | 航天飞机图像 | 112 | 5.5×1010 | 物理地址 |
之前的工作已经用PCR扩增时需要的引物作为唯一的地址或标识符标记DNA序列[40, 41],当检索特定文件时,可以利用该文件的唯一标识符仅通过PCR放大该文件相关的DNA序列[14]。但是,基于DNA的数据访问流程需要在2个方面进行改进,即多次访问和索引。首先,由于在进行文件读取时需要使用PCR等DNA富集技术,多次访问数据会从根本上破坏原始数据的完整性。其次,需要适当的索引方法来支持DNA数据存储的海量数据管理。为了从海量数据存储中有选择地检索特定的文件,应该为每个文件赋予名称和大小等元数据,并对元数据进行频繁访问,根据元数据信息确定目标DNA文库对其进行测序解码,而不是频繁访问整个数据库。
目前已经开发了适用于DNA数据存储的微流控读取设备、二维码存储文件元数据以及调节PCR特异性扩增检索文件元数据等多种DNA数字信息检索方式。但是,为了进一步向DNA数据存储的工业应用迈进,仍需要开发一种适合DNA数据存储访问过程的数据管理系统。
2.1 数字微流控设备检索数字微流控设备是一种可精确控制和操纵微尺度流体的技术,尤其特指亚微米结构的技术[42]。在对存储有数字信息的DNA进行保存时,通常希望以尽量小的物理空间实现对存储不同文件的DNA的高效分隔。而微流控设备非常适合对这种极小的物理空间中的DNA进行精确操纵,可以自动化的形式获取目标位置的DNA,从而方便对其进行后期的扩增以及测序等操作。
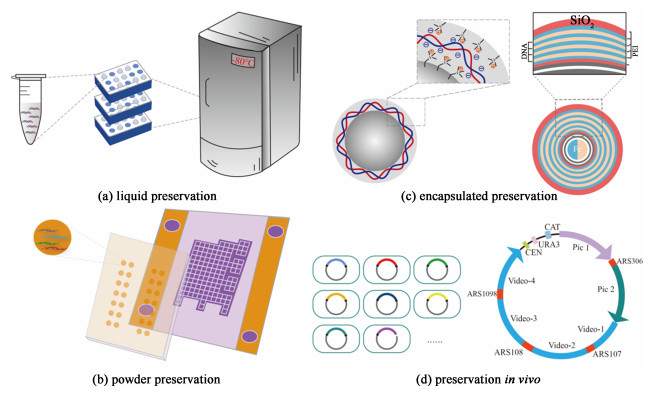
2019年华盛顿大学的Ceze团队[43]将50个大小为1 151 μm的DNA斑点分别分隔在玻璃板表面大小为2.7 mm×2.7 mm的50个格子中,实现了对DNA的高效分隔,同时为每个DNA斑点赋予了不同的物理地址,可用于高信息密度的长期存储。而通过微流控设备可高效获取目标位置的DNA斑点,不同的斑点中的DNA文库可以使用相同的引物,并且这些斑点可以密集的排列在微流体装置上,而不会对目标文件的读取造成明显的污染,避免了之前利用引物作为地址对文件进行读取时不同引物之间的扩增特异性问题,每个玻璃板可存储50 TB数据,单个斑点可以存储高达1 TB的数据。2022年,Antkowiak等[44]通过将包封有不同文件的二氧化硅球固定在玻璃板的不同格子中,对文件形成了物理隔离,之后配合微流控设备,实现了对目标文件的选择性访问,大大延长了这个DNA数据存储系统中DNA的保存时间(图 2)。
|
| 图 2 二氧化硅与微流控设备结合保存DNA Fig.2 Silica combined with microfluidic devices to preserve DNA |
| |
元数据,又称中介数据,为描述数据的数据,主要是描述数据属性的信息。为了选择性的访问海量的数据中的特定文件,通常需要频繁的读取与每个文件的内容相关的元数据来搜索数据库,而不是频繁的读取整个数据库,这样可以大大节约测序成本及解码需要的计算资源。因此,为元数据设计一个可频繁读取的数据存储介质,对于文件的选择性访问也具有很重要的意义。
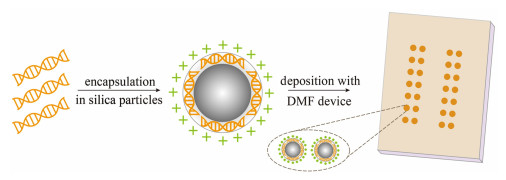
二维码是按照一定规律分布在平面上的、黑白相间的、能够记录数据符号信息的图案[45],它利用多个与二进制相对应的几何图形来表示文字数值信息,可通过摄像头等成像设备提取存储在其中的数据,在频繁读取方面具有非常大的优势。Choi等[46]开发了一种具有二维码图案的DNA微盘,首先将DNA文库中存储的文件的名称、大小以及扩增所需的引物信息作为元数据存储进二维码图案中,之后把DNA文库连接到已经固化为此二维码图案的聚合物上。此DNA微盘既具有可频繁访问的光学索引,又可以直接访问数据编码的DNA分子,与现有的基于DNA微阵列的DNA管理系统相比,该研究通过将DNA固定在微盘上,在防止不同DNA文库之间的污染方面具有额外的优势,并且DNA微盘尺寸较小,也可以对存储不同文件的DNA文库进行高效的物理分隔,对于提高DNA的物理存储密度也具有很大的优势[图 3(a)]。
|
| 图 3 元数据检索 Fig.3 Metadata retrieval |
| |
理想状态下,每个二氧化硅粒子中包封的DNA中存储的文件应该是不同的,并且进行文件访问时,应当能够快捷的只获取存有目标文件的粒子,只对存有目标文件的粒子进行DNA释放及扩增测序,这样才能实现文件的高效读取。但是,由于包封有DNA的二氧化硅或碱金属盐离子的直径在25~200 nm之间,粒子极小,因此,在快速获取存储目标文件的粒子方面仍存在挑战。2021年,Banal等[47]通过给存储不同文件的二氧化硅球做特征标记,实现了目标文件的选择性访问。他们将存储有不同文件的DNA分别包封在不同的二氧化硅球中,并在二氧化硅表面固定3条代表文件特征的DNA序列;在进行文件访问时,加入与这3条DNA序列互补的修饰有荧光基团的DNA序列,即可通过荧光信号分选出目标粒子,对其进行数据访问[图 3(b)]。
2.3 PCR扩增检索在通过PCR扩增以及测序技术对DNA溶液中的文件进行读取时,通常会得到包含所有文件的DNA测序数据,对所有文件进行解码,这大大增加了测序以及解码的成本。面对大规模数据存储时,此种方式显然是不切实际的。为了对DNA溶液中的文件进行选择性读取,2018年,Organick等[14]对35个总计大小超过200 MB的文件进行编码,将其存放在同一个DNA文库中,并为每个文件设计了唯一的PCR引物序列,利用特定的引物对存储有35个文件的DNA溶液进行PCR扩增,实现了对于目标文件的选择性读取[图 4(a)]。

|
| 图 4 PCR扩增 Fig.4 PCR amplification |
| |
PCR扩增过程中分子间相互作用的特异性是热力学可调的。通过控制PCR的扩增特异性也可以实现元数据的访问。Tomek等[48]将PCR引物设计为与DNA序列之间的汉明距离(Hamming distance,HD)分别为0、2、4和6,通过调节PCR过程中的退火温度、引物浓度、盐离子浓度等反应条件,控制PCR的反应特异性。当反应特异性最强时,仅能扩增出0HD的DNA序列,对这些序列进行测序解码,可获得所存储文件的大致轮廓,实现对文件的预览。而随着扩增的非特异性逐渐增强,可对实现整个文库中DNA序列的扩增,读取出整个文件[图 4(b)]。文件的预览功能也是元数据检索的一种形式,可以大大降低测序成本。
2.4 细胞体内信息检索当大规模数据存储至细胞体内时,数据的读取方式仍面临挑战。Qi等[39]以混菌培养形式在质粒中存储的445 KB数字信息,在进行文件访问时,利用限制性内切酶Not Ⅰ获取DNA文库,对酶切产物进行二代测序,当测序读长的覆盖深度达到1000×时才能实现完美解码,这大大增加了测序成本。2020年,Chen等[38]将254 KB的数据写入一条人工染色体中在进行文件读取时,需要将酵母体内的天然染色体与人工染色体全部进行测序,增加了测序成本;并且将来源于天然染色体与人工染色体的测序读长区分开,实现了人工染色体的基因组从头组装在生物信息学领域也是一大挑战。
3 DNA信息存储设备集成化研究进展尽管关于DNA的保存研究已经非常广泛和全面,但是对于现有的保存方式与工业应用的兼容性和实用性、面对海量数据文库的集成的可操作性,数据的稳健性与安全性还没有系统性的评估。通常为了方便对DNA文件进行读取,需将存储有不同文件的DNA序列物理分隔开。而面对大规模数据存储,目前的承载介质将占用极大的物理空间,从而明显降低DNA的物理存储密度。本论文对现有的4种承载介质在面对海量数据存储时的检索方式、操作难度以及集成的难易程度做了比较(表 2),希望为能适应大规模数据存储的新型DNA承载介质的开发提供参考,以对DNA进行高效的物理分隔,提高DNA的物理存储密度,实现目标文件的便捷读取。
| 承载介质 | 检索方式 | 操作难度 | 集成难度 |
| 液状 | 核酸扩增 | 容易 | 能耗较大、占用空间较大 |
| 干粉 | 数字微流控 | 容易 | DNA储存时间较短、占用空间较大 |
| 二氧化硅/碱金属盐 | 数字微流控、布尔逻辑 | 困难 | 成本较高 |
| 细胞 | 酶切、核酸扩增 | 容易 | 数据存储总量小、占用空间较大 |
虽然目前的方法大多有自动化合成[19]和测序设备的帮助(例如柱合成、阵列合成、Illumina、纳米孔测序仪等)[13, 14, 21, 22],这些仪器对于生物学的研究具有非常重要的意义,但是他们并非是为数据存储而设计的,并且许多中间步骤仍然需要专业的实验室技术人员来执行。首先,需要通过高通量设备合成DNA文库,之后将合成的DNA文库保存在物理介质中,最后取出保存的DNA文库放入DNA测序仪中进行测序[49-51]。目前的方法中每个步骤都会涉及液体操作和手动操作,这些限制了技术的放大能力使其难以应用于大规模存储,并且增加了错误的概率,为了纠正这些错误就会需要大量的数据冗余,这又限制了数据存储的效率。除此之外,数据的随机访问可能涉及将地址标签添加到DNA序列中,随着规模的扩大,这可能会变得非常麻烦[52]。由于扩增的偏差和偏好性,使用PCR随机访问数据也可能导致数据的丢失[9, 53]。因此,即使使用自动流体系统,开发基于DNA的完全集成、高效和实用的数据库仍然具有挑战性。如果DNA数据存储的写入、存储及整个流程没有实现完全的集成自动化,那么未来很难应对海量数据存储。
华盛顿大学[54]开发了一种自动化的端到端的DNA数据存储设备(图 5),该设备系统有3个核心模块,分别是编码/解码软件模块、DNA合成模块、DNA制备和测序模块,编码软件模块负责翻译和在有效负载序列中添加错误校正,将数据编码成DNA序列,然后DNA合成模块使用定制的DNA合成器将其写入DNA寡核苷酸,一旦合成完成,合成的DNA将被洗脱到一个存储容器中,DNA被汇集并进行液体存储,接着,DNA制备和测序模块中的注射泵将制备的混合物分配到存储容器中,用纳米孔测序仪进行读取。虽然在21 h内存储5 Byte还不具备商业可行性,但是之后的研究对数量级进行了改进。该集成设备将各模块最大化,可以重复使用,降低了整个系统的复杂性,平衡了设置和运行模块所需的成本和人工投入。
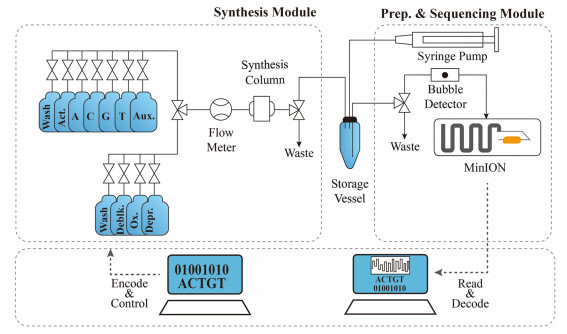
|
| 图 5 端到端的DNA数据存储设备 Fig.5 End-to-end DNA data storage device |
| |
Xu等[55]报道了一个完全集成的基于电化学DNA合成和单电极测序的可扩展的DNA数据存储系统(图 6)。在该系统中,二进制数据首先使用ReedSolomon(RS码)转换为DNA序列,DNA是在Au电极上基于亚磷酰胺化学和电化学脱保护进行合成,测序是通过在同一电极上使用合成测序的方法完成的,由聚合酶催化的引物延伸引起电荷重新分布,从而形成可测量的电流尖峰。用于合成和测序的所有液体操作都是基于一种SlipChip的滑动芯片装置完成的,通过该装置,涉及复杂的DNA合成和测序过程的液体操作可以大大的简化。测序后的电极再生,可以实现重复测序,提高精度,且使用电极阵列几乎可以同时并行完成DNA合成和测序,这也可以显著增加未来给定时间内存储的数据量。电极通过Au NP修饰后可以在表面合成高密度的DNA分子,从而增强测序过程中电荷信号的再分布,另外DNA序列不需要地址序列,有利于开发高度集成的微型化数据存储装置,通过扩大微芯片上电极和反应库的数量,该方法可能用于开发一个真正大型的DNA数据库,具有高效率、集成化和自动化的未来。
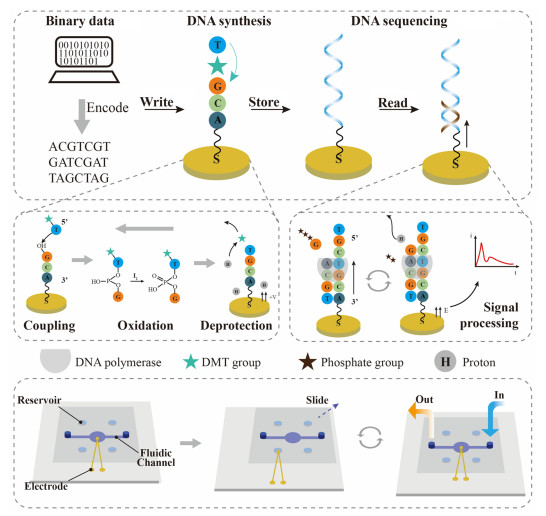
|
| 图 6 基于电化学DNA合成和单电极测序的可扩展的DNA数据存储系统 Fig.6 Scalable DNA data storage system based on electrochemical DNA synthesis and single-electrode sequencing |
| |
尽管大量研究证明,DNA的储存容量、可扩展性、稳定性等方面优于现有的硅基设备存储技术,但是由于读写过程复杂,写、存、读3个步骤难以集成化。目前的DNA数据存储技术中,已经通过低温冷冻、脱水干燥、二氧化硅或碱金属盐包封以及细胞体内保存等方式实现了DNA的长期稳定保存,并且已经通过数字微流控设备实现了对于具有物理地址的特定DNA文库进行自动化读取,另外,还通过二维码以及PCR特异性扩增等方式实现了数据的选择性检索。然而,现有的DNA保存介质都只适用于实验室小规模的数据存储,在面临海量数据存储时,仍然存在以下问题:(1)物理存储密度较低,目前的研究的存储密度要远远低于DNA的理论存储密度;(2)从大规模集成化应用的角度方面看,虽然DNA合成成本有所下降,但是相比于DNA测序成本,依然跟不上其发展的步伐,且随着合成长度的增加,碱基合成的错误率也在不断地增加;(3)针对目标文件难以进行便捷的选择性读取,现有的研究都存在读取速度较慢的不足,面对大规模数据的存储,如何实现数据的物理隔离,保证对大型数据库进行无错误的访问也是一个亟需解决的问题,且能源消耗也是一个非常重要的因素;(4)难以将DNA的合成、保存及测序步骤集成化,开发一种集成度高、效率高、实用性强的DNA数据存储设备仍然是一个挑战,实现DNA数据存储的全自动化仍是一个亟待解决的问题。
希望将来可以开发出新型的自动化集成设备,并且结合能够对DNA进行高效的物理分隔的新型的DNA承载介质,配合元数据检索方式,实现对于特定文件的低成本高效读取。我们相信随着生物及信息技术的不断发展,这些问题都将得到解决,使DNA数据存储技术能够进入到商业应用中。
| [1] |
REINSEL D, GANTZ J, RYDNING J. The digitization of the world from edge to core[R]. Framinham: International Data Corporation, 2018
|
| [2] |
DE SILVA P Y, GANEGODA G U. New trends of digital data storage in DNA[J]. BioMed Research International, 2016, 2016: 8072463. |
| [3] |
BONNET J, COLOTTE M, COUDY D, et al. Chain and conformation stability of solid-state DNA: Implications for room temperature storage[J]. Nucleic Acids Research, 2010, 38(5): 1531-1546. DOI:10.1093/nar/gkp1060 |
| [4] |
AKRAM F, HAQ I U, ALI H, et al. Trends to store digital data in DNA: An overview[J]. Molecular Biology Reports, 2018, 45(5): 1479-1490. DOI:10.1007/s11033-018-4280-y |
| [5] |
CARMEAN D, CEZE L, SEELIG G, et al. DNA data storage and hybrid molecular-electronic computing[J]. Proceedings of the IEEE, 2019, 107(1): 63-72. DOI:10.1109/JPROC.2018.2875386 |
| [6] |
EXTANCE A. How DNA could store all the world's data[J]. Nature, 2016, 537: 22-24. DOI:10.1038/537022a |
| [7] |
LEE H H, KALHOR R, GOELA N, et al. Terminator-free template-independent enzymatic DNA synthesis for digital information storage[J]. Nature Communications, 2019, 10: 2383. DOI:10.1038/s41467-019-10258-1 |
| [8] |
KNYPHAUSEN P, LINDENBURG L, HOLLFELDER F. Error-free synthetic DNA by molecular dictation[J]. Trends in Biotechnology, 2021, 39(9): 861-865. DOI:10.1016/j.tibtech.2021.02.001 |
| [9] |
CHEN Y, TAKAHASHI C N, ORGANICK L, et al. Quantifying molecular bias in DNA data storage[J]. Nature Communications, 2020, 11: 3264. DOI:10.1038/s41467-020-16958-3 |
| [10] |
ANTKOWIAK P L, LIETARD J, DARESTANI M Z, et al. Low cost DNA data storage using photolithographic synthesis and advanced information reconstruction and error correction[J]. Nature Communications, 2020, 11: 5345. DOI:10.1038/s41467-020-19148-3 |
| [11] |
XIONG A, YAO Q, PENG R, et al. PCR-based accurate synthesis of long DNA sequences[J]. Nature Protocols, 2006, 1: 791-797. DOI:10.1038/nprot.2006.103 |
| [12] |
GAO X, LEPROUST E, ZHANG H, et al. A flexible light-directed DNA chip synthesis gated by deprotection using solution photogenerated acids[J]. Nucleic Acids Research, 2001, 29(22): 4744-4750. DOI:10.1093/nar/29.22.4744 |
| [13] |
ERLICH Y, ZIELINSKI D. DNA Fountain enables a robust and efficient storage architecture[J]. Science, 2017, 355(6328): 950-954. DOI:10.1126/science.aaj2038 |
| [14] |
ORGANICK L, ANG S D, CHEN Y J, et al. Random access in large-scale DNA data storage[J]. Nature Biotechnology, 2018, 36: 242-248. DOI:10.1038/nbt.4079 |
| [15] |
GRASS R N, HECKEL R, PUDDU M, et al. Robust chemical preservation of digital information on DNA in silica with error-correcting codes[J]. Angewandte Chemie (International Ed in English), 2015, 54(8): 2552-2555. DOI:10.1002/anie.201411378 |
| [16] |
郜艳敏, 唐梦童, 刘倩, 等. DNA信息存储中关键生化方法的研究[J]. 合成生物学, 2021, 2(3): 384-398. GAO Yanmin, TANG Mengtong, LIU Qian, et al. The pivotal biochemical methods in DNA data storage[J]. Synthetic Biology Journal, 2021, 2(3): 384-398. (in Chinese) |
| [17] |
ANCHORDOQUY T J, MOLINA M C. Preservation of dna[J]. Cell Preservation Technology, 2007, 5(4): 180-188. DOI:10.1089/cpt.2007.0511 |
| [18] |
GAO Y, CHEN X, QIAO H, et al. Low-bias manipulation of DNA oligo pool for robust data storage[J]. ACS Synthetic Biology, 2020, 9(12): 3344-3352. DOI:10.1021/acssynbio.0c00419 |
| [19] |
KOSURI S, CHURCH G M. Large-scale de novo DNA synthesis: Technologies and applications[J]. Nature Methods, 2014, 11: 499-507. DOI:10.1038/nmeth.2918 |
| [20] |
CARUTHERS M H. The chemical synthesis of DNA/RNA: Our gift to science[J]. The Journal of Biological Chemistry, 2013, 288(2): 1420-1427. DOI:10.1074/jbc.X112.442855 |
| [21] |
CHURCH G M, GAO Y, KOSURI S. Next-generation digital information storage in DNA[J]. Science, 2012, 337(6102): 1628. DOI:10.1126/science.1226355 |
| [22] |
HOSSEIN T Y S M, GABRYS R, MILENKOVIC O. Portable and error-free DNA-based data storage[J]. Scientific Reports, 2017, 7: 5011. DOI:10.1038/s41598-017-05188-1 |
| [23] |
LINDAHL T. Instability and decay of the primary structure of DNA[J]. Nature, 1993, 362(6422): 709-715. DOI:10.1038/362709a0 |
| [24] |
Richa, SINHA R P, HÄDER D P. Physiological aspects of UV-excitation of DNA[J]. Topics in Current Chemistry, 2015, 356: 203-248. |
| [25] |
LINDAHL T, NYBERG B. Rate of depurination of native deoxyribonucleic acid[J]. Biochemistry, 1972, 11(19): 3610-3618. DOI:10.1021/bi00769a018 |
| [26] |
LINDAHL T, KARLSTRÖM O. Heat-induced depyrimidination of deoxyribonucleic acid in neutral solution[J]. Biochemistry, 1973, 12(25): 5151-5154. DOI:10.1021/bi00749a020 |
| [27] |
SHAPIRO R, KLEIN R S. The deamination of cytidine and cytosine by acidic buffer solutions. Mutagenic implications[J]. Biochemistry, 1966, 5(7): 2358-2362. DOI:10.1021/bi00871a026 |
| [28] |
SHAPIRO R, DANZIG M. Acidic hydrolysis of deoxycytidine and deoxyuridine derivatives. The general mechanism of deoxyribonucleoside hydrolysis[J]. Biochemistry, 1972, 11(1): 23-29. DOI:10.1021/bi00751a005 |
| [29] |
MIDDAUGH C R, EVANS R K, MONTGOMERY D L, et al. Analysis of plasmid DNA from a pharmaceutical perspective[J]. Journal of Pharmaceutical Sciences, 1998, 87(2): 130-146. DOI:10.1021/js970367a |
| [30] |
FUCIARELLI A F, WEGHER B J, BLAKELY W F, et al. Yields of radiation-induced base products in DNA: Effects of DNA conformation and gassing conditions[J]. International Journal of Radiation Biology, 1990, 58(3): 397-415. DOI:10.1080/09553009014551761 |
| [31] |
ZOLTEWICZ J A, CLARK D F, SHARPLESS T W, et al. Kinetics and mechanism of the acid-catalyzed hydrolysis of some purine nucleosides[J]. Journal of the American Chemical Society, 1970, 92(6): 1741-1749. DOI:10.1021/ja00709a055 |
| [32] |
CHEN W, KOHLL A X, NGUYEN B H, et al. Combining data longevity with high storage capacity—Layer-by-layer DNA encapsulated in magnetic nanoparticles[J]. Advanced Functional Materials, 2019, 29(28): 1901672. DOI:10.1002/adfm.201901672 |
| [33] |
KOHLL A X, ANTKOWIAK P L, CHEN W, et al. Stabilizing synthetic DNA for long-term data storage with earth alkaline salts[J]. Chemical Communications, 2020, 56(25): 3613-3616. DOI:10.1039/D0CC00222D |
| [34] |
WONG P C, WONG K K, FOOTE H. Organic data memory using the DNA approach[J]. Communications of the ACM, 2003, 46(1): 95-98. DOI:10.1145/602421.602426 |
| [35] |
YACHIE N, SEKIYAMA K, SUGAHARA J, et al. Alignment-based approach for durable data storage into living organisms[J]. Biotechnology Progress, 2007, 23(2): 501-505. DOI:10.1021/bp060261y |
| [36] |
AILENBERG M, ROTSTEIN O. An improved Huffman coding method for archiving text, images, and music characters in DNA[J]. BioTechniques, 2009, 47(3): 747-754. DOI:10.2144/000113218 |
| [37] |
BANCROFT C, BOWLER T, BLOOM B, et al. Long-term storage of information in DNA[J]. Science, 2001, 293(5536): 1763-1765. |
| [38] |
CHEN W, HAN M, ZHOU J, et al. An artificial chromosome for data storage[J]. National Science Review, 2021, 8(5): nwab028. DOI:10.1093/nsr/nwab028 |
| [39] |
HAO M, QIAO H, GAO Y, et al. A mixed culture of bacterial cells enables an economic DNA storage on a large scale[J]. Communications Biology, 2020, 3: 416. DOI:10.1038/s42003-020-01141-7 |
| [40] |
BORNHOLT J, LOPEZ R, CARMEAN D M, et al. A DNA-based archival storage system[C]//Proceedings of the Twenty-First International Conference on Architectural Support for Programming Languages and Operating Systems. Atlanta, Georgia, USA: ACM, 2016: 637-649
|
| [41] |
HOSSEIN TABATABAEI YAZDI S M, YUAN Y, MA J, et al. A rewritable, random-access DNA-based storage system[J]. Scientific Reports, 2015, 5: 14138. DOI:10.1038/srep14138 |
| [42] |
CHOI K, NG A H C, FOBEL R, et al. Digital microfluidics[J]. Annual Review of Analytical Chemistry, 2012, 5: 413-440. DOI:10.1146/annurev-anchem-062011-143028 |
| [43] |
NEWMAN S, STEPHENSON A P, WILLSEY M, et al. High density DNA data storage library via dehydration with digital microfluidic retrieval[J]. Nature Communications, 2019, 10: 1706. DOI:10.1038/s41467-019-09517-y |
| [44] |
ANTKOWIAK P L, KOCH J, NGUYEN B H, et al. Integrating DNA encapsulates and digital microfluidics for automated data storage in DNA[J]. Small, 2022, 18(15): e2107381. DOI:10.1002/smll.202107381 |
| [45] |
杨军, 刘艳, 杜彦蕊. 关于二维码的研究和应用[J]. 应用科技, 2002, 29(11): 11-13. YANG Jun, LIU Yan, DU Yanrui. The study and application of the two-dimensional code[J]. Applied Science and Technology, 2002, 29(11): 11-13. (in Chinese) |
| [46] |
CHOI Y, BAE H J, LEE A C, et al. DNA micro-disks for the management of DNA-based data storage with index and write-once-read-many (WORM) memory features[J]. Advanced Materials, 2020, 32(37): e2001249. DOI:10.1002/adma.202001249 |
| [47] |
BANAL J L, SHEPHERD T R, BERLEANT J, et al. Random access DNA memory using Boolean search in an archival file storage system[J]. Nature Materials, 2021, 20: 1272-1280. DOI:10.1038/s41563-021-01021-3 |
| [48] |
TOMEK K J, VOLKEL K, INDERMAUR E W, et al. Promiscuous molecules for smarter file operations in DNA-based data storage[J]. Nature Communications, 2021, 12: 3518. DOI:10.1038/s41467-021-23669-w |
| [49] |
SANGER F, NICKLEN S, COULSON A R. DNA sequencing with chain-terminating inhibitors[J]. Proceedings of the National Academy of Sciences of the United States of America, 1977, 74(12): 5463-5467. |
| [50] |
ESCALONA M, ROCHA S, POSADA D. A comparison of tools for the simulation of genomic next-generation sequencing data[J]. Nature Reviews Genetics, 2016, 17: 459-469. DOI:10.1038/nrg.2016.57 |
| [51] |
ROTHBERG J M, HINZ W, REARICK T M, et al. An integrated semiconductor device enabling non-optical genome sequencing[J]. Nature, 2011, 475: 348-352. DOI:10.1038/nature10242 |
| [52] |
TOMEK K J, VOLKEL K, SIMPSON A, et al. Driving the scalability of DNA-based information storage systems[J]. ACS Synthetic Biology, 2019, 8(6): 1241-1248. DOI:10.1021/acssynbio.9b00100 |
| [53] |
KEBSCHULL J M, ZADOR A M. Sources of PCR-induced distortions in high-throughput sequencing data sets[J]. Nucleic Acids Research, 2015, 43(21): e143. |
| [54] |
TAKAHASHI C N, NGUYEN B H, STRAUSS K, et al. Demonstration of end-to-end automation of DNA data storage[J]. Scientific Reports, 2019, 9: 4998. DOI:10.1038/s41598-019-41228-8 |
| [55] |
XU C, MA B, GAO Z, et al. Electrochemical DNA synthesis and sequencing on a single electrode with scalability for integrated data storage[J]. Science Advances, 2021, 7(46): eabk0100. DOI:10.1126/sciadv.abk0100 |
 2025, Vol. 42
2025, Vol. 42