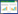维生素D3是人体生长与维持所必需的重要微量元素之一[1],其代谢产物1, 25-二羟维生素D3是人体内激素的活性形式,参与调节多种代谢反应[2]。人体中的维生素D3主要来自于2个方面:一是直接从食物中获得,一是皮肤接受紫外线照射而以7-脱氢胆甾醇为底物合成[3]。7-脱氢胆甾醇可以在无细胞体系中通过光化学反应生成维生素D3[4],因此合成7-脱氢胆甾醇是生产维生素D3的关键步骤。

|
| 图 1 人体内维生素D3的摄取与代谢 Figure 1 Vitamin D3 intake and metabolism in human body |
| |
目前,合成7-脱氢胆甾醇的技术主要分为化学合成[5]和生物转化[6],化学合成副产物多、条件难控制、环境污染严重。生物转化原料成本较高、酶来源相对有限,都无法适应大规模生产。合成生物学的兴起,为重要医药产品的合成与清洁能源的生产提供了理论基础和技术保证,为变革医药生产方式和转变能源供应模式提供了无限的空间和可能[7]。在工程学思想地指导下,结合代谢工程的基础理论,将基因元件有机组织起来,构成功能模块或生物网络。选择适宜的底盘生物,通过对微生物固有代谢路径的改造和利用,协调外源基因的表达与内源代谢路径的平衡,赋予生物细胞以崭新的生命功能,实现多种医药产品[8-9]和新型能源[10-11]的异源合成。“元件、模块、底盘”这一统一的整体,必须协调运作,才能实现目标产物的高效合成。
作为合成生物学的基础,底盘细胞的选择至关重要,它承载着所有人工设计的生物零件,并且它的稳定性必须足以保证复杂的生物系统正常运转并发挥功能[12]。酿酒酵母是一种常见的工业微生物,被广泛应用于生产各种各样的次级代谢物。同时,因为酿酒酵母是首个被全基因组测序的真核生物,它也成为分子生物学研究中的典型的模式生物。基于对酿酒酵母基因信息的了解,大量分子生物学的工具被开发,使得异源基因在酿酒酵母中得以稳定的表达[13-14]。解脂酵母作为产油脂模式酵母菌,能利用多种碳水化合物和脂肪为碳源进行生长,对于甘油酯和烷烃类均有较强的代谢能力,这使得它非常适合于工业生产的应用。根据合成生物学的指导思想,通过引入人源的C24还原酶基因dhcr24,构建能够异源合成7-脱氢胆甾醇的工程酵母[15]。通过比较生长、产量、进一步改造的潜力,分析两种底盘对于异源表达7-脱氢胆甾醇的优劣。
1 材料与方法 1.1 菌株、质粒、培养基本研究所使用的大肠杆菌(Escherichia coli)BMDH5α购买于博迈德公司,酿酒酵母(Saccharomyces cerevisiae)BY4742以及在此基础上敲除erg5基因的单基因缺失株ΔERG5均由本实验室保存,解脂酵母(Yarrowia lipolytica)野生型CIBTS0949和缺陷型CIBTS0996菌株由上海工业技术研发中心杨晟老师馈赠。
| 菌株 | 基因型 |
| BY4742 | MATα his3Δ1 leu2Δ0 lys2Δ0 ura3Δ0 |
| ΔERG5 | MATα his3Δ1 leu2Δ0 lys2Δ0 ura3Δ0 erg5Δ::KanMX |
| CIBTS0949 | 购买于Agricultural Research Service Culture Collection, USA |
| CIBTS0996 | matA, ura3-302, leu2-270, lys8-11 |
本研究所使用的大肠杆菌-酿酒酵母穿梭质粒pRS425为本实验室保存。
使用LB培养基对大肠杆菌进行培养和保藏,具体成分为:胰蛋白胨10 g·L-1,酵母提取物5 g·L-1,NaCl 10 g·L-1;固体培养基添加2%的琼脂粉。筛选大肠杆菌转化子时向LB培养基中加入100 μg·mL-1的氨苄青霉素。酵母菌发酵使用YPD培养基,具体成分为:酵母提取物10 g·L-1,细菌蛋白胨20 g·L-1,葡萄糖20 g·L-1。重组酵母菌株筛选用SC drop-out培养基,具体成分为:酵母氮源6.7 g·L-1,氨基酸缺省粉末混合物2 g·L-1,葡萄糖20 g·L-1;固体培养基添加2%的琼脂粉。
1.2 基因元件的获得与菌株构建C24还原酶基因dhcr24编码一个依赖于FAD的氧化还原酶,催化甾体类物质24位双键的还原[16]。酿酒酵母与解脂酵母的密码子偏好性相差较大,根据Genbank中人源C24还原酶的氨基酸序列(GI:114155130),分别根据酿酒酵母与解脂酵母的密码子偏好性进行优化,由金唯智公司全合成该基因。
以酿酒酵母BY4742的基因组为模版,PCR获得启动子ENO2p和终止子ADH1t,用重叠延伸PCR的方法将启动子、基因和终止子拼接成1个大片段,用限制性内切酶PstⅠ和BamHⅠ消化该片段,之后连接至用相同的酶消化后的载体pRS425上,得到pSW01,测序保证其序列的正确性。将pSW01分别导入酿酒酵母BY4742和ΔERG5中,得到SyBE_Sc01100001和SyBE_Sc01100002。
克隆含有解脂酵母TEF启动子、Lip2t终止子以及200bp非解脂酵母内源序列t1、t2的质粒pYL(t1-TEFp-LIP2t-t2),启动子和终止子之间有两个背向BsmBⅠ酶切位点,可使人源的dhcr24基因无痕连入启动子和终止子之间,得到C24基因表达盒t1-TEFp-dhcr24-LIP2t-t2。200 bp非解脂酵母内源序列t1和t2的作用是为避免因为与基因组其他区域有较强的同源性而发生错误整合。由于解脂酵母中尚未发现天然质粒可供用于蛋白表达,我们将C24基因表达盒整合到解脂酵母中rDNA位点。以解脂酵母CIBTS0949的基因组为模版,扩增获得整合位点上游片段rDNA(up)、下游片段rDNA(down)和筛选标记URA,从质粒pYL上扩增获得200 bp非解脂酵母内源序列t1和t2,用重叠延伸PCR和酶切连接的方式获得rDNA(up)-URA-t1片段和t2-rDNA(down)片段。将3个大片段同时导入解脂酵母中,利用同源重组将dhcr24基因表达盒整合到rDNA簇上(图 2),通过对转化子进行菌落PCR验证,得到SyBE_YL01100001~SyBE_YL01100004。

|
| 图 2 解脂酵母中dhcr24基因整合策略 Figure 2 Genome integration for dhcr24 in Y.lipolytica |
| |
| 引物 | 序列(5′ to 3′) |
| ENO2p-F(PstI) | ctgcaggtgtcgacgctgcgggtata |
| ENO2p-DHCR24-R | acaacaaagcgcagactgccaaagagacagcaggttccattattattgtatgttatagta |
| DHCR24-ENO2p-F | tcataacaccaagcaactaatactataacatacaataataatggaacctgctgtctcttt |
| DHCR24-ADH1t-R | tggagacttgaccaaacctctggcgaagaagtccaaagcttcaatgtctagctgccttac |
| ADH1t-DHCR24-F | tgaggtttacgacaagatttgtaaggcagctagacattgaagctttggacttcttcgcca |
| ADH1t-R(BamHI) | ggatcccatgccggtagaggtgtggt |
| BsaI-Ura new-F | ggtctcacgcccagagagccattga |
| BsaI-Ura new-R | ggtctcaagaccaaaggcctgtttctcggtgt |
| rDNA(up)-F | gcggccgcgctcgatcctaaggggtgg |
| rDNA(up)-R | tcagactgagaccggtctcaggcgtggctaccttaagagagtcatagtt |
| BsaI-t1-F | ggtagccacgccagagaccggtctcagtctgaagaatgaatgatttgatga |
| BsaI-t1-R | taataatactcctcaaattgctacc |
| t2-F | gtgaatttactttaaatcttgca |
| t2-R | taattagatgacgaggcattatccacaatgtatcaggtatcta |
| rDNA(down)-F | ctgatacattgtggataatgcctcgtcatctaattagtgac |
| rDNA(down)-R | gcggccgccttcggtat |
| c-t1-F | gtctgaagaatgaatgatttgatga |
| c-t2-R | atccacaatgtatcaggtatcta |
| 注:酶切位点用斜体+下划线表示和同源臂用下划线标出。 | |
采用醋酸锂法进行酵母转化[17],具体步骤如下:1) 挑取酵母单菌落,接入4 mL适宜的培养基中,30 ℃过夜培养。2) 取适量菌液转接到新鲜培养基中,初始OD为0.1,30 ℃培养5 h,OD达到0.5左右。3)4 000 r/min离心5 min收集菌体,无菌水洗2遍,0.1 mol/L醋酸锂洗1遍,置于冰上,得到感受态细胞。4)99.9 ℃煮ssDNA 10 min,迅速置于冰上静置5 min。5) 准备转化体系,见表 3。6) 将转化体系加入感受态中,涡旋混匀,30 ℃孵育30 min。7)42 ℃水浴中热击20 min。8)4 000 r/min离心5 min,丢弃上层清液,用无菌水洗1遍。9) 用100 μL无菌水重悬菌体,涂在适宜的SC drop-out固体培养基上。30 ℃培养2~4 d。
| 成分 | 体积/μL |
| DNA片段 | 1 |
| ssDNA | 10.0 |
| 50% PEG3350 | 240 |
| 1M LiAc | 36 |
| ddH2O | 补至360 |
挑取酵母单菌落,接入4 mL培养基中,30 ℃ 220 r/min过夜培养,OD600达到4左右,转接至50 mL相应的培养基中,继续培养24 h。取二级种子接入100 mL YPD发酵培养基中,初始OD600为0.2,30 ℃、220 r/min摇床振荡培养。
1.5 产物提取方法[18]5 000 r/min离心5 min,丢弃上层清液,收集菌体,用无菌水洗掉细胞表面的培养基成分。液氮冷冻并充分研磨,将细胞粉末加入干净的离心管中,称取细胞湿重。向研磨后的细胞中加入5 mL 1.5 mol/L KOH-甲醇溶液,80 ℃皂化90 min。加入2 mL正己烷,涡旋10 min萃取产物。5 000 r/min离心2 min,将上层清液转移至干净的离心管中,冷冻干燥约30 min。用400 μL正己烷溶解产物,再次冻干(大于4 h)。加入200 μL衍生化试剂MSTFA, 37 ℃反应2 h。
1.6 GC-TOF-MS检测方法[19]用气相色谱-飞行时间质谱(GC-TOF-MS)对产物进行定性和定量分析。
分析条件如下:电离方式为电子轰击电离EI+,电子束能量为70 eV,离子化电流为40 μA。质谱扫描范围在m/z 50~800,离子源温度为250 ℃,进样口温度为260 ℃,氦气(99.9995%)作为载气,91 kPa恒压模式下操作。柱温的变化情况为:首先在70 ℃维持2 min,之后以30 ℃·min-1的速度升至250 ℃,再以10 ℃·min-1的速度升到280 ℃并维持15 min,最后以5 ℃·min-1的速度升到290 ℃,并维持5 min。

|
| 图 3 7-脱氢胆甾醇标准曲线 Figure 3 Standard curve of 7-dehydrocholesterol |
| |
将含有dhcr24基因的质粒pSW01导入酿酒酵母BY4742菌株中得到重组酵母菌株SyBE_Sc01100001,以野生型酿酒酵母BY4742作为对照,同时进行摇瓶发酵,以考察引入dhcr24基因后,菌体的生长以及7-脱氢胆甾醇的合成情况。用紫外可见分光光度计测量发酵液在不同发酵时间于波长600 nm处的吸光度,得到菌株的生长曲线。如图 4所示,SyBE_Sc01100001和野生型BY472,生长趋势上无明显差别。当发酵进行到10~12 h时,细胞进入一个短暂的平台期,随后进入二次生长,60 h后进入平台期,最终生物量OD600达到19左右(图 4)。

|
| 图 4 酿酒酵母菌株生长曲线 Figure 4 Growth curves for S.cerevisiae |
| |
当SyBE_Sc01100001和对照组BY4742同时进入稳定期后(72 h),离心收集细胞,提取酵母细胞中的总固醇,并用GC-TOF-MS对产物进行分析。7-脱氢胆甾醇标准品的分析结果表明,其出峰时间为20.83 min,质谱的碎片有325、351和366 3个特征峰。但是SyBE_Sc01100001的GC-TOF-MS分析结果并没有保留时间为20.83 min的峰,而是与BY4742的结果几乎完全一致,在21.46 min出现1个较高的峰,通过质谱分析,该物质为麦角固醇(图 5)。这表明,在酿酒酵母中,麦角固醇合成路径制约了7-脱氢胆甾醇的合成。

|
| 图 5 酿酒酵母菌株GC-TOF-MS分析结果 Figure 5 GC-TOF-MS analysis for S.cerevisiae |
| |
为了实现在酿酒酵母中合成7-脱氢胆甾醇,内源的麦角固醇合成路径需要被抑制或阻断。我们选择了缺失erg5基因的BY4742菌株(ΔERG5) 作为底盘,将含有dhcr24基因的质粒pSW01导入后,获得了1株重组酵母SyBE_Sc01100002。从生长曲线上来看,缺失erg5基因的ΔERG5菌株在对数期的生长情况与BY4742类似,但是12 h之后没有二次生长。人源的dhcr24基因对ΔERG5的生长没有产生明显的影响(图 4)。由于7-脱氢胆甾醇不会降解亦不会被酿酒酵母利用,为了便于与BY4742和SyBE_Sc01100001菌株比较,在分析ΔERG5和SyBE_Sc01100002的产物时,选择72 h作为发酵终点。SyBE_Sc01100002在保留时间为20.82 min的峰为7-脱氢胆甾醇的峰,摇瓶发酵SyBE_Sc01100002得到的7-脱氢胆甾醇的产量为2.62 mg·L-1。ΔERG5和SyBE_Sc01100002在22.58 min都有1个较高的峰,通过质谱分析,该物质为fecosterol,也是麦角固醇合成路径上的中间体,为酵母固醇被ERG6催化所得的产物(图 5)。
2.2 解脂酵母底盘中7-脱氢胆甾醇的合成为了寻找更适于生产7-脱氢胆甾醇的底盘酵母,将dhcr24基因整合到解脂酵母CIBTS0996基因组的rDNA簇位点上,得到SyBE_YL01100001~SyBE_YL01100004 4株工程酵母,同时进行发酵,每隔3 h取样测定OD600,得到生长曲线(图 6)。0~6 h为延滞期,随后进入对数生长期,18 h后几乎同时进入平台期。虽然同为dhcr24整合菌株,但是彼此之间的生长有差异,SyBE_YL01100002最终生物量OD600只有5左右,SyBE_YL01100001略高有7左右,SyBE_YL01100003和SyBE_YL01100004最终OD600达到8左右。

|
| 图 6 解脂酵母菌株生长曲线 Figure 6 Growth curves for Y.lipolytica |
| |
发酵24 h后收集全部细胞,用GC-TOF-MS对产物进行定性和定量分析(图 7)。解脂酵母中,保留时间为20.82 min的峰为7-脱氢胆甾醇的峰,21.46 min的峰为麦角固醇的峰,这说明麦角固醇合成途径没有影响dhcr24在解脂酵母中的表达。在同样的整合策略下获得的几株能够生产7-脱氢胆甾醇的解脂酵母菌株彼此生长有差异,产物组成相同,但是目标产物7-脱氢胆甾醇的产量有明显差异,对应于菌株SyBE_YL01100001~SyBE_YL01100004的摇瓶发酵产量分别是19.04、16.97、27.91和25.89 mg·L-1。

|
| 图 7 解脂酵母GC-TOF-MS分析结果 Figure 7 GC-TOF-MS analysis for Y.lipolytica |
| |
1) 为了解除微生物自身代谢物对外源产物的竞争性抑制,需要对微生物天然合成路径进行改造,而改造的难度与该代谢物对微生物生长的重要性有直接的关系。7-脱氢胆甾醇的合成与酵母内源麦角固醇的合成拥有许多相似的步骤。酵母中麦角固醇的合成是一个多酶参与的复杂过程[20],根据不同酶编码的基因对酵母生长和代谢的影响程度可以将麦角固醇合成途径上的基因分为必需基因和非必需基因[21]。从酵母固醇到麦角固醇,需要ERG6、ERG2、ERG3、ERG5和ERG4这5个酶的催化,编码这5个酶的基因均为非必需基因[22]。敲除erg5基因能缓解麦角固醇对7-脱氢胆甾醇的竞争性抑制,但是ΔERG5菌株的生物量远低于野生型BY4742,这表明麦角固醇的缺失对酿酒酵母的生长产生显著的影响。通过发酵优化改善麦角固醇缺失菌株的生长状况,有利于7-脱氢胆甾醇的合成。副产物fecosterol的出现说明可以通过敲除其他非必需基因而进一步抑制麦角固醇合成路径,避免有限的碳源被浪费。
2) 在内源的代谢路径未被改造的情况下,通过基因整合首次实现了在解脂酵母中生产7-脱氢胆甾醇,其生物量与酿酒酵母ΔERG5菌株相当,但是产量具有绝对的优势。解脂酵母细胞内有油滴,油滴的大小和数量明显大于酿酒酵母。解脂酵母中使用不同的碳源时油滴的大小会发生变化:采用葡萄糖和甘油等普通碳源时,显微镜下观测不到明显的油滴,但是采用油脂类的物质作为碳源时,油滴的数量多且大小明显[23]。油滴是由中性脂质构成的,所以对于一些疏水性或者极性较弱的物质溶解性好,在生产甾醇类物质的时候会减小细胞的负担。由于在解脂酵母中rDNA簇进行基因整合属于多位点整合[24],所以外源基因的拷贝数不同可能是几株7-脱氢胆甾醇生产菌株生长与产量存在差异的原因。
3) 任何与次级代谢相关的基因改造都会对7-脱氢胆甾醇的产量造成影响。例如,过表达甲羟戊酸途径的相关基因可以提高7-脱氢胆甾醇的前体供应,发现酵母内源合成路径的关键节点,从而有目的地进行底盘菌株的改造。酿酒酵母遗传背景清晰,适用于酿酒酵母的遗传操作工具常见易得,未来进一步的基因改造的空间巨大。虽然整合策略带来了较高的产量,但是适用于解脂酵母的分子操作工具有限,可以开发更多的工具,使得对解脂酵母的分子操作变得简单易行。例如,除本研究使用的TEF启动子之外,EXP、GPD、GPAT、YAT、XPR2和FBA都是解脂酵母中常见的启动子[25]。
4) 解脂酵母具有酿酒酵母所不具备的多种碳源的利用能力,大分子被细胞分泌的甲壳素打散成小分子颗粒,进入解脂酵母内。脂类先被解脂酵母分泌出的脂肪酶分解为甘油三磷酸和脂肪酸,甘油三磷酸转化为甘油三酰,脂肪酸一方面经过β氧化最终转化成乙酰辅酶A,另一方面也会为甘油三酰的合成提供底物。烷烃类物质被打散进入细胞后变成脂肪醇,进而被氧化成为脂肪酸,通过β氧化最终转化为乙酰辅酶A[26-27],解脂酵母对于甾醇类物质生产而言具有广泛的应用前景。
| [1] | Holick M F. Vitamin D: Importance in the prevention of cancers, type 1 diabetes, heart disease, and osteoporosis[J]. The American Journal of Clinical Nutrition, 2004, 79: 362–371. |
| [2] | Bendik I, Friedel A, Roos F F, et al. Vitamin D: A critical and essential micronutrient for human health[J]. Frontiers in Physiology, 2014, 5: 248–248. |
| [3] | Carlberg C. Genome-wide (over)view on the actions of vitamin D[J]. Frontiers in Physiology, 2014, 5: 167–167. |
| [4] | Lehmann U, Hirche F, Stangl G I, et al. Bioavailability of vitamin D2 and D3 in healthy volunteers, a randomized placebo-controlled trial[J]. Journal of Clinical Endocrinology & Metabolism, 2013, 98: 4339–4345. |
| [5] | Confalone P N, Kulesha I D, Uskokovic M R. A new synthesis of 7-dehydrocholesterol[J]. The Journal of Organic Chemistry, 1981, 46: 1030–1032. DOI: 10.1021/jo00318a041 |
| [6] | Lewis D, Galczenski H, Needle S, et al. Enzyme inhibition during the conversion of squalene to cholesterol[J]. Steroids, 1995, 60: 475–483. DOI: 10.1016/0039-128X(95)00054-T |
| [7] |
刘夺, 杜瑾, 赵广荣, 等. 合成生物学在医药及能源领域的应用[J].
化工学报, 2011, 62: 2391–2397.
Liu Duo, Du Jin, Zhao Guangrong, et al. The application of synthetic biology in medicine and energy[J]. CIESC Journal, 2011, 62: 2391–2397. DOI: 10.3969/j.issn.0438-1157.2011.09.002 |
| [8] | Ro D K, Paradise E M, Ouellet M, et al. Production of the antimalarial drug precursor artemisinic acid in engineered yeast[J]. Nature, 2006, 440: 940–943. DOI: 10.1038/nature04640 |
| [9] | Ajikumar P K, Xiao W, Tyo K E J, et al. Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli[J]. Science, 2010, 330: 70–74. DOI: 10.1126/science.1191652 |
| [10] | Li H, Cann A F, Liao J C. Biofuels: Biomolecular engineering fundamentals and advances[J]. Annual Review of Chemical and Biomolecular Engineering, 2010, 1: 19–36. DOI: 10.1146/annurev-chembioeng-073009-100938 |
| [11] | Steen E J, Kang Y, Bokinsky G, et al. Microbial production of fatty-acid-derived fuels and chemicals from plant biomass[J]. Nature, 2010, 463: 559–562. DOI: 10.1038/nature08721 |
| [12] |
赵学明, 王庆昭. 合成生物学:学科基础、研究进展与前景展望[J].
前沿科学, 2007, 3: 56–66.
Zhao Xueming, Wang Qingzhao. Synthetic biology: Subject basis, research development and prospect forecast[J]. Frontier Science, 2007, 3: 56–66. DOI: 10.3969/j.issn.1673-8128.2007.03.010 |
| [13] | Siddiqui M S, Thodey K, Trenchard I, et al. Advancing secondary metabolite biosynthesis in yeast with synthetic biology tools[J]. FEMS Yeast Research, 2012, 12: 144–170. DOI: 10.1111/j.1567-1364.2011.00774.x |
| [14] |
刘夺, 张莹, 周晓, 等. 合成生物技术生产甾体激素中间体的研究展望[J].
生命科学, 2013, 25: 958–965.
Liu Duo, Zhang Ying, Zhou Xiao, et al. The research forecast of the production of steroid hormone intermediates by synthetic biology[J]. Chinese Bulletin of Life Science, 2013, 25: 958–965. |
| [15] | Groenewald M, Boekhout T, Neuvéglise C, et al. Yarrowia lipolytica: Safety assessment of an oleaginous yeast with a great industrial potential[J]. Critical Reviews in Microbiology, 2014, 40(3): 187–206. DOI: 10.3109/1040841X.2013.770386 |
| [16] | Lang C. Preparation of 7-dehydrocholesterol and/or the biosynthetic intermediates and/or secondary products thereof in transgenic organisms: US, 2011/0151509 A1[P]. 2011-07-23 |
| [17] | Gietz R D, Schiestl R H. High-efficiency yeast transformation using the LiAc/SS carrier DNA/PEG method[J]. Nature Protocol, 2007, 2: 31–34. DOI: 10.1038/nprot.2007.13 |
| [18] | 周晓. 反复间歇真空发酵过程中酿酒酵母适应性进化的系统分析[D]. 天津: 天津大学, 2011 Zhou Xiao. Systematic investigation on adaptive evolution of Saccharomyces cerevisiae to repeated-batch vacuum fermentation[D]. Tianjin: Tianjin University, 2011(in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10056-1012022095.htm |
| [19] | 丁明珠. 酵母对纤维素水解液中复合抑制剂耐受的系统分析与解耦[D]. 天津: 天津大学, 2011 Ding Mingzhu. Systematic analysis and decoupling of yeast tolerance to combined inhibitors in lignocellulose hydrolysates[D]. Tianjin: Tianjin University, 2011(in Chinese) http://cdmd.cnki.com.cn/Article/CDMD-10056-1012007508.htm |
| [20] | Dupont S, Lemetais G, Ferreira T, et al. Ergosterol biosynthesis: A fungal pathway for life on land[J]. Evolution, 2012, 66: 2961–2968. DOI: 10.1111/evo.2012.66.issue-9 |
| [21] |
林晓珊, 江宏文, 张毅. 酵母麦角固醇生物合成及其基因调控的研究[J].
生物学杂志, 2010, 27: 83–86.
Lin Xiaoshan, Jiang Hongwen, Zhang Yi. The research of yeast ergosterol biosynthesis and relevant genetic regulation[J]. Journal of Biology, 2010, 27: 83–86. DOI: 10.3969/j.issn.1008-9632.2010.02.083 |
| [22] | Kristan K, Rižner T L. Steroid-transforming enzymes in fungi[J]. The Journal of Steroid Biochemistry and Molecular Biology, 2012, 129: 79–91. DOI: 10.1016/j.jsbmb.2011.08.012 |
| [23] | Athenstaedt K, Jolivet P, Boulard, et al. Lipid particle composition of the yeast Yarrowia lipolytica depends on the carbon source[J]. Proteomics, 2006, 6(5): 1450–1459. DOI: 10.1002/(ISSN)1615-9861 |
| [24] |
赵鹤云, 黄瑛, 杨江科, 等. 解脂耶氏酵母表达系统研究进展[J].
生物加工过程, 2008, 6(3): 10–16.
Zhao Heyun, Huang Ying, Yang Jingke, et al. The research on the expression system of Yarrowia lipolytica[J]. Chinese Journal of Bioprocess Engineering, 2008, 6(3): 10–16. |
| [25] | John B, Leqian L, Heidi R, et al. Tuning gene expression in Yarrowia lipolytica by a hybrid promoter approach[J]. Applied and Environmental Microbiology, 2011, 77(22): 7905–7914. DOI: 10.1128/AEM.05763-11 |
| [26] | Mlíččková K, Roux E, Athenstaedt K, et al. Lipid accumulation, lipid body formation, and acyl coenzyme A oxidases of the yeast Yarrowia lipolytica[J]. Applied and Environmental Microbiology, 2004, 70(7): 3918–3924. DOI: 10.1128/AEM.70.7.3918-3924.2004 |
| [27] | Beopoulos A, Mrozova Z, Thevenieau F, et al. Control of lipid accumulation in the yeast Yarrowia lipolytica[J]. Applied and Environmental Microbiology, 2008, 74(24): 7779–7789. DOI: 10.1128/AEM.01412-08 |
 2017, Vol. 34
2017, Vol. 34