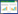核黄素(维生素B2)是生物合成黄素单核苷酸(FMN)和黄素腺嘌呤二核苷酸(FAD)的重要前体物,广泛用于饲料、食品、医药及化妆品等行业[1, 2]。核黄素的工业生产方法主要有化学合成法和微生物发酵法。微生物发酵法以其成本低廉、环境污较小、生产周期短等优势成为目前核黄素生产的主流方法。
目前,核黄素的主要生产菌株有棉阿舒囊霉(Ashbya gossypii)、假丝酵母(Candida famata)和枯草芽孢杆菌(B. subtilis)[3]。由于这些菌种主要是通过多轮理化诱变之后筛选得到,经过传统诱变得到的菌株会产生大量不利突变,同时由于随机突变的不确定性使得通过代谢工程进一步合理改进这些工业菌株面临着极大的挑战和困难[4]。大肠杆菌(E. coli)因其生长速度快、基因操作技术成熟、生物合成途径以及酶的研究透彻等特征,已经被广泛用于氨基酸、核苷酸、生物塑料以及其它专用化学品的工业化生产,例如:丝氨酸、苏氨酸、色氨酸,肌苷、鸟苷、PHA、乳酸、琥珀酸和乙醇等[5]。
原核生物合成核黄素的代谢途径如图 1所示:前体物三磷酸鸟苷(GTP)和5-磷酸核酮糖以1:2的比例,经过GTP咪唑环的开环水解、脱氨基、还原、脱磷酸、C4单位生成、二氧四氢蝶啶合成以及核黄素合成七步酶促反应,最终转化为核黄素[6]。在B. subtilis中,核黄素生物合成相关酶的编码基因在染色体上以操纵子的形式存在。如图 2所示[7],该操纵子位于染色体的210°,总长度4.3 kb,由ribG、ribB、ribA、ribH、ribT 5个基因和5′端的RFN结构(ribO)组成。此外,在RFN结构的上游有一个一级启动子ribP1,ribB基因的末端和ribT基因的上游分别有两个二级启动子ribP2和ribP3。核黄素操纵子转录受到FMN/FAD的反馈调节[7]。大肠杆菌ribF基因编码产物为核黄素激酶和黄素腺嘌呤二核苷酸氨酰转移酶,这两个酶在枯草芽孢杆菌中是由ribC编码,该基因的突变能够解除枯草芽孢杆菌核黄素操纵子的转录调控,提高核黄素的积累[8](图 1)。

|
| 图 1 大肠杆菌和枯草芽孢杆菌核黄素生物合成途径 Fig.1 Riboflavin biosynthesis is in prokaryotes. The E. coli genes encoding each enzymatic activity are indicated on the left while the corresponding B. subtilis genes are indicated to the right. Abbreviations: Ru5P, ribulose-5-phosphate; GTP, guanosine triphosphate; DARPP, 2,5-diamino-6-ribosylamino-4(3H)-pyrimidinone- 5’-phosphate; ARPP, 5-amino-6-(5’-phosphoribo sylamino) uracil; ArPP, 5-amino-6-(5’-phosphoribitylamino)uracil; ArP, 4-(1-D-ribitylamino)-5-amino-2,6-dihydroxypyrimidine; DHPB, 3,4-dihydroxy-2-butanone 4-phosph ate; DRL, 6,7-dimethyl-8-ribityl-lumazine; FMN, flavin mono- nucleotide; FAD, flavin adenine dinucleotide |

|
| 图 2 枯草芽孢杆菌rib操纵子结构 Fig.2 Structure of the rib operon from B. subtilis. The bold arrows show the mutual arrangement and the direction of transcription for the five rib operon genes: ribG, ribB, ribA, ribH and ribT. The fine arrows denote the direction of transcription from the P1, P2 and P3 promoters in the rib operon. The hairpin structures indicate the locations of ρ-independent transcription terminators |
本论文针对大肠杆菌中的核黄素生物合成途径进行改造,使其来生产核黄素,主要包括枯草芽孢杆菌rib操纵子的异源表达和ribF基因的减弱表达。首先在E. coli MG1655中以质粒形式表达B. subtilis 168核黄素合成的rib操纵子。该rib操纵子由强启动子Ptrc介导,同时删除了rib操纵子5′端RFN元件和ribO调控序列,解除了rib操纵子转录水平的反馈抑制,并在ribG上游加入人工设计的核糖体结合位点(RBS)序列。通过酶切连接到不同拷贝数的质粒,并对诱导剂IPTG浓度和发酵温度进行了优化;在此最优条件下减弱表达E. coli本身的ribF基因,最终的菌株ECX4在LB培养基中核黄素产量提高到292.3 mg·L-1,为构建大肠杆菌核黄素生产菌株及其进一步改造奠定了基础。
1 实验部分 1.1 主要试剂和实验仪器试剂:氨苄青霉素、奇霉素、四环素、酵母抽提物、胰蛋白胨、基因组提取试剂盒、DNA胶回收试剂盒、PCR产物纯化试剂盒和质粒DNA小量抽提试剂盒均购自上海生工生物工程有限公司;Fast Pfu高保真快速DNA聚合酶购自北京全式金,限制性内切酶和T4 DNA连接酶均购自加拿大Fermentas公司。其他试剂为国产分析纯。
仪器:TU1901双光束紫外可见分光光度计(北京普析通用仪器有限公司);SBA40C生物传感仪(山东省科学院生物研究所);超声波破碎仪(美国Sonics & Materials公司)。
1.2 菌株和质粒菌株与质粒见表 1。
| Strain or plasmid | Relevant characteristics | Source |
| Strains | ||
| B.subtili s 168 | Cloning rib operon | Lab |
| E.coli DH5α | Plasmid construcion | Lab |
| MG1655 | E. coli K-12 MG1655, wild-type | CGSC * |
| ECS1 | MG1655, containing pSC101 | This study |
| ECS2 | MG1655, containing p15A | This study |
| ECS3 | MG1655, containing pBR322 | This study |
| ECX1 | MG1655, containing pSC101-BSrib | This study |
| ECX2 | MG1655, containing p15A-BSrib | This study |
| ECX3 | MG1655, containing pBR322-BSrib | This study |
| ECX4 | ECX3, ribF-pJ23115 | This study |
| Plasmids | ||
| pTKRED | Spc r,pSC101 replication,temperature sensitive replication origin,P araBAD-driven I-SceI gene,recombinase expression plasmid,lac-inducible | [9] |
| pTKS/CS | Cm r,Tet r,p15A replication,I-SceI restriction sites | [9] |
| pSC101 | Amp r, pSC101 replication, Ptrc-driven, expression vector | Lab |
| p15A | Amp r, p15A replication, Ptrc-driven, expression vector | Lab |
| pBR322 | Amp r, pBR322 replication, Ptrc-driven, expression vector | Lab |
| pSC101-BSrib | Amp r, pSC101 replication, Ptrc-synRBS- ribD-ribE-ribA-ribH | This study |
| p15A-BSrib | Amp r, p15A replication, Ptrc-synRBS- ribD-ribE-ribA-ribH | This study |
| pBR322-BSrib | Amp r, pBR322 replication, Ptrc-synRBS- ribD-ribE-ribA-ribH | This study |
| *Coli Genetic Stock Center. | ||
引物设计见表 2。
| Primer name | Sequence 5′→3′ |
| rib1 | GCGCGAATTCTACTAGAGATTAAAGAGGAGAAAATGGAAGAGTATTATATGAAGCTGGCC |
| rib2 | CGCGGATCCTTATTCAAATGAGCGGTTTAAATTTG |
| ribF P1 | CTTTGTCGCCAGCTTCGAT |
| ribF P2 | CCCTACATTGTACCAAGGGCTGAGCTAGCTATAAAGAAGAAACTGACCGCCTGCT |
| ribF P3 | TTTATAGCTAGCTCAGCCCTTGGTACAATGTAGGGATAACAGGGTAATGTACC |
| ribF P4 | GCTAGCATTGTACCAAGGGCTGAGCTAGCTATAAAATTACCCTGTTATCCCTACTAAG |
| ribF P5 | TTATAGCTAGCTCAGCCCTTGGTACAATGCTAGCTGAGCCAGACATGAAGCTGAT |
| ribF P6 | TTACGCGCAAATGCTTCACC |
| tetA1 | GCGTGAAGTGGTTCGGTTG |
| tetA2 | CTGTAATGCAGGTAAAGCGATC |
首先用试剂盒提取枯草芽孢杆菌基因组DNA并以其为模板,用引物rib1/rib2进行BSrib操纵子的PCR扩增。纯化后的BSrib片段与载体pSC101分别经EcoRI和BamHI双酶切,酶切产物纯化后用T4 DNA连接酶连接得到表达载体pSC101-BSrib。表达载体p15A-BSrib和pBR322-BSrib以相同方式构建。将这3个质粒电转化到E. coli DH5α感受态细胞,涂布含有氨苄青霉素的LB平板,挑选抗性克隆,提质粒进行EcoRI和BamHI双酶切鉴定。
1.5 菌株的构建ECS1、ECS2、ECS3菌株(阴性对照)的构建:将空质粒pSC101、p15A、pBR322分别电转化至大肠杆菌MG1655感受态中,涂布于含有100 μg·mL-1氨苄青霉素的LB平板,37 ℃过夜培养,筛选阳性转化子。
ECX1、ECX2、ECX3菌株的构建:将构建好的质粒pSC101-BSrib、p15A-BSrib、pBR322-BSrib分别电转化至大肠杆菌MG1655感受态中,涂布于含有100 μg·mL-1氨苄青霉素的LB平板,37 ℃过夜培养,筛选阳性转化子。
ECX4菌株的构建:ribF片段构建参照文献[10]。将构建好的ribF片段电转化至含有pTKRED质粒的大肠杆菌ECX3感受态中,涂布于含有100 μg·mL-1奇霉素、10 μg·mL-1四环素、100 μg·mL-1氨苄青霉素和2 mmol·L-1 IPTG的LB平板,30 ℃过夜培养,挑选抗性克隆进行PCR验证。挑取抗性单克隆于5 mL含有100 μg·mL-1奇霉素、100 μg·mL-1氨苄青霉素、2 mmol·L-1 IPTG和0.2% L-阿拉伯糖的LB的试管中,30 ℃、220 r/min过夜培养。0.2% L-阿拉伯糖用于诱导I-SceI核酸内切酶表达,删除四环素抗性基因tetA。第2天,稀释涂布于含有100 μg·mL-1奇霉素、100 μg·mL-1氨苄青霉素、2 mmol·L-1 IPTG和0.2% L-阿拉伯糖的LB平板,30 ℃过夜培养,挑选阳性克隆进行PCR验证。
1.6 培养基LB培养基:10 g·L-1胰蛋白胨,5 g·L-1酵母抽提物,10 g·L-1氯化钠,121 ℃高压蒸汽灭菌20 min。LB发酵培养基:10 g·L-1胰蛋白胨,5 g·L-1酵母抽提物,10 g·L-1氯化钠,20.9 g·L-1 3-(N-吗啉基)丙磺酸,用1 mol·L-1 NaOH调节pH值到7.4,115 ℃高压蒸汽灭菌30 min。
1.7 摇瓶发酵培养挑取单菌落于装有5 mL LB培养基的试管中,37 ℃、220 r/min过夜培养。以初始OD600约为0.01转接到50 mL LB中进行种子培养,当菌体处于对数生长期时接入到100 mL LB (500 mL摇瓶)发酵培养基中,使初始OD600达到0.01左右,分别添加葡萄糖、氨苄青霉素至浓度为10 g·L-1、100 μg·mL-1,220 r/min振荡培养。
1.8 代谢产物分析细胞生长是用紫外可见分光光度计于波长600 nm处测定吸光度值。核黄素是用紫外可见分光光度计于波长444 nm处测定吸光度值,由公式Riboflavin titer=(OD444-0.0057)×稀释倍数/0.0321 计算得到。培养基中的葡萄糖用生物传感仪检测。
1.9 核黄素激酶/黄素腺嘌呤二核苷酸氨酰转移酶酶活测定在添加10 g·L-1葡萄糖的LB培养基中培养菌体至对数生长期时收集菌液,12 000×g、4 ℃离心10 min,收集菌体;用2 mL的0.1 mmol·L-1 pH 7.5磷酸缓冲液溶液(4 ℃预冷)洗涤2次,悬浮在2 mL同样的缓冲液中,进一步用超声波破碎菌体;4 ℃条件下,13 000×g离心10 min去除菌体等杂质,得上清即为粗酶液,放置冰上备用。核黄素激酶/黄素腺嘌呤二核苷酸氨酰转移酶活性测定参照Bandyopadhyay[11]方法。蛋白浓度测定参照Bradford[12]方法,以牛血清蛋白作为参照。
2 结果与讨论 2.1 rib操纵子的克隆将rib操纵子上游的调控序列删除之后同时也将ribG的RBS序列也删除,因此在引物rib1中加入合成的RBS序列。然后以B. subtilis 168基因组DNA为模板,以rib1/rib2为引物,PCR扩增得到B. subtilis染色体上的核黄素操纵子基因(BSrib)片段。经琼脂糖凝胶电泳,结果表明,在大小约3.5 kb处有目的条带,与理论值相同(图 3a)。将扩增得到的BSrib片段用EcoRI和BamHI双酶切,并分别和表达载体pSC101、p15A和pBR322连接后得到最终质粒pSC101-BSrib、p15A-BSrib和pBR322-Bsrib。对以上3个表达质粒经EcoRI和BamHI酶切鉴定,经琼脂糖凝胶电泳,分别在3.5和4.9 kb,3.5和3.9 kb,3.5和4.1 kb有条带,与预期结果一致(图 3b)。对pSC101-BSrib、p15A-BSrib和pBR322-BSrib质粒中的rib操纵子测序后发现与NCBI公布的核酸序列完全一致,表明质粒构建成功。

|
| 图 3 核黄素操纵子BSrib表达质粒的构建及验证 Fig.3 Construction and identification of BSrib operon expression plasmid. a) BSrib fragment produced by PCR. b) Identification of expression plasmid digested with EcoRI and BamHI. 1: BSrib PCR fragment; 2: pSC101-BSrib; 3: p15A-BSrib; 4: pBR322-Bsrib; M: 1 kb DNA ladder |
将过表达枯草芽孢杆菌核黄素操纵子的重组质粒pSC101-BSrib(低拷贝)、p15A-BSrib(中等拷贝)和pBR322-BSrib(高拷贝)分别电转化到菌株E. coli MG1655中得到菌株ECX1、ECX2和ECX3。为测定不同质粒拷贝数对核黄素产量的影响,在37 ℃、2 mmol·L-1 IPTG浓度条件下对其进行摇瓶发酵实验。如表 3所示,随质粒拷贝数的增加,核黄素产量大幅度提高。与低拷贝质粒pSC101-BSrib相比,高拷贝质粒pBR322-BSrib的核黄素产量和得率分别增加了1.8和1.3倍。同时,将带有空质粒pSC101、p15A和pBR322的阴性对照菌株ECS1、ECS2和ECS3在相同条件下发酵,结果并未检测到有核黄素生成。
| Production Strains | OD 600 | Riboflavin titer/(mg·L -1) | Riboflavin yield/(mg·g -1) |
| ECX1 | 5.9±0.1 | 43.2±2.3 | 19.2±1.1 |
| ECX2 | 6.8±0.1 | 84.5±2.2 | 32.9±1.2 |
| ECX3 | 7.2±0.1 | 121.6±2.7 | 44.2±0.9 |
发酵过程中很多参数会影响核黄素的生产,包括诱导剂IPTG浓度、温度等。其中诱导剂IPTG浓度是影响核黄素发酵生产的重要因素。IPTG浓度过高,很大程度上会抑制菌体生长,造成核黄素产量降低。IPTG浓度过低则导致核黄素操纵子的表达不充分,也会影响核黄素产量。为了增加核黄素的积累,优化发酵条件,探讨了不同的IPTG浓度对菌株ECX3发酵的影响。
如表 4所示,研究了0.05、0.10、0.25、0.50、1.00和2.00 mmol·L-1 IPTG浓度下菌株ECX3在37 ℃时生产核黄素的状况。结果表明0.10 mmol·L-1为最适宜浓度,在此条件下核黄素产量为233.2 mg·L-1。与2.00 mmol·L-1 IPTG浓度相比,产量提高91.8%。
| IPTG concentration/(mmol·L -1) | OD 600 | Riboflavin titer/(mg·L -1) | Riboflavin yield/(mg·g -1) |
| 0.05 | 8.8±0.3 | 208.5±15.3 | 62.2±4.2 |
| 0.10 | 8.1±0.3 | 233.2±3.5 | 76.2±3.7 |
| 0.25 | 7.8±0.2 | 186.3±4.7 | 59.8±1.5 |
| 0.50 | 7.5±0.1 | 135.4±6.1 | 47.3±1.9 |
| 1.00 | 7.3±0.1 | 126.8±2.2 | 45.6±0.1 |
| 2.00 | 7.2±0.1 | 121.6±2.7 | 44.2±0.9 |
温度是影响微生物生长和调节生命活动的重要因素,严格保持菌体的生长繁殖和生长合成所需的最适温度,对稳定发酵过程、缩短发酵周期、提高发酵产量有一定的现实意义。
在上述最适宜IPTG浓度条件下,分别考察了菌株ECX3在33、35、37、42和44 ℃ 5个温度下的核黄素生成情况,结果见表 5。结果表明,在一定温度范围内,随温度的升高,核黄素产量呈升高趋势。温度超过42 ℃以后,核黄素产量下降。42 ℃为最适宜温度,在此条件下核黄素产量最高,为251.4 mg·L-1。这可能是因为GTP环水解酶II是核黄素生物合成的限速酶,其最适作用温度为42 ℃[13]。
| Temperature/℃ | OD 600 | Riboflavin titer/(mg·L -1) | Riboflavin yield/(mg·g -1) |
| 33 | 8.1±0.1 | 183.7±27.8 | 59.7±9.3 |
| 35 | 8.6±0.2 | 203.4±8.5 | 62.0±2.4 |
| 37 | 8.1±0.3 | 233.2±3.5 | 76.2±3.7 |
| 42 | 9.8±0.8 | 251.4±9.0 | 77.7±2.9 |
| 44 | 8.3±0.1 | 208.9±19.5 | 63.3±6.6 |
为了减弱工程菌株ECX3核黄素激酶/黄素腺嘌呤二核苷酸氨酰转移酶(ribF)的表达以降低FMN和FAD的合成,并提高核黄素的积累。将构建好的ribF片段电转化到重组感受态细胞并涂布在四环素平板,30 ℃过夜培养,挑选抗性克隆进行PCR验证。其琼脂糖凝胶电泳结果如图 4a)所示,在约2 kb处有目的条带,符合预期结果。四环素抗性基因tetA被删除后,挑选阳性克隆进行PCR验证。其琼脂糖凝胶电泳结果如图 4b)所示,在789 bp处有目的条带,2 kb处没有条带,证明四环素抗性基因tetA已经被删除,并测序验证。ribF无痕操作成功,基因组上原有的ribF启动子和调控序列替换为J23115启动子,ECX4菌株的构建成功。

|
| 图 4 ECX4工程菌株的构建 Fig.4 Construction of the engineering strain ECX4 by λ-Red recombination. a) Insertion of tetA for replacing the promoter ribF.b) Deletion of tetA gene. M: 1 kb DNA ladder |
首先从酶学特性考察菌株ECX4中核黄素激酶/黄素腺嘌呤二核苷酸氨酰转移酶的表达效果,对ECX3和ECX4的核黄素激酶/黄素腺嘌呤二核苷酸氨酰转移酶酶活进行了测定。菌株ECX4核黄素激酶/黄素腺嘌呤二核苷酸氨酰转移酶比酶活为1.02±0.06 nmol·(min mg-protein)-1,而对应的野生型菌株为1.58±0.10 nmol·(min mg-protein)-1,重组菌的比酶活降低了35.4%。
为测定菌株ECX4生产核黄素的能力,在0.1 mmol·L-1 IPTG、42 ℃条件下对重组菌株ECX4和出发菌株ECX3进行发酵实验。摇瓶发酵结果显示菌株ECX4生产了292.3 mg·L-1核黄素,比野生菌提高了16.3%。Mack等[8]发现在B. subtilis中突变核黄素激酶/黄素腺嘌呤二核苷酸氨酰转移酶,能够显著降低该酶的酶活,从而提高核黄素的产量。
3 结论1)本研究以大肠杆菌E. coli K-12 MG1655出发菌株,以质粒的形式过量表达枯草芽孢杆菌B. subtilis的rib操纵子,实现了核黄素的积累,并且核黄素合成能力随着质粒拷贝数的增加而增强。然后对诱导剂IPTG浓度和发酵温度进行了优化。摇瓶发酵结果显示,0.1 mmol·L-1 IPTG和42 ℃为核黄素生产的最佳条件。该条件下菌株ECX3的核黄素产量达到251.4 mg·L-1。
2)通过无痕基因操作技术,减弱工程菌株ECX3核黄素激酶/黄素腺嘌呤二核苷酸氨酰转移酶(ribF)的表达,得到菌株ECX4。摇瓶发酵核黄素产量达到292.3 mg·L-1,与ECX3相比,产量提高了16.3%。在后续的研究中,通过分子生物学手段进一步改造,以降低副产物、提高核黄素产量,这对大肠杆菌产核黄素的研究具有重要的意义。
| [1] | Massey V. The chemical and biological versatility of riboflavin[J]. Biochem Soc Trans, 2000, 28(4): 283-296 |
| [2] | Koizumi S, Yonetani Y, Maruyama A, et al. Production of riboflavin by metabolically engineered Corynebacterium ammonia genes[J]. Appl Microbiol Biotechnol, 2000, 53(6): 674-679 |
| [3] | Stahmann K P, Revuelta J L, Seulberger H. Three biotechnical processes using Ashbya gossypii, Candida famata, or Bacillus subtilis compete with chemical riboflavin production[J]. Appl Microbiol Biotechnol, 2000, 53(5): 509-516 |
| [4] | Peberdy J F. Biology of industrial microorganisms[C]//Demain NAS Ed.. Biology of Penicillins. Menlo Park: Benjamin-Cummings, 1985 |
| [5] | Zhang F, Rodriguez S, Keasling J D. Metabolic engineering of microbial pathways for advanced biofuels production[J]. Curr Opin Biotechnol, 2011, 22(6): 775-783 |
| [6] | Bacher A, Eberhardt S, Fischer M, et al. Biosynthesis of vitamin b2 (riboflavin)[J]. Annu Rev Nutr, 2000, 20: 153-167 |
| [7] | Sklyarova S A, Kreneva R A, Perumov D A, et al. The characterization of internal promoters in the Bacillus subtilis riboflavin biosynthesis operon[J]. Genetika, 2012, 48(10): 967-974 |
| [8] | Mack M, van Loon A P, Hohmann H P. Regulation of riboflavin biosynthesis in Bacillus subtilis is affected by the activity of the flavokinase/flavin adenine dinucleotide synthetase encoded by ribC[J]. J Bacteriol, 1998, 180(4): 950-955 |
| [9] | Kuhlman T E, Cox E C. Site-Specific chromosomal integration of large synthetic constructs[J]. Nucleic Acids Res, 2010, 38(6): e92 |
| [10] | Goswami R S. Targeted gene replacement in fungi using a split-marker approach[J]. Methods Mol Biol, 2012, 835: 255-269 |
| [11] | Bandyopadhyay D, Chatterjee A K, Datta A G. Effect of cadmium, mercury and copper on partially purified hepatic flavokinase of rat[J]. Mol Cell Biochem, 1997,167(1/2): 73-80 |
| [12] | Bradford M M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Anal Biochem, 1976, 72: 248-254 |
| [13] | Hümbelin M, Griesser V, Keller T, et al. GTP cyclohydrolase II and 3,4-dihydroxy-2-butanone 4-phosphate synthase are rate-limiting enzymes in riboflavin synthesis of an industrial Bacillus subtilis strain used for riboflavin production[J]. J Ind Microbiol Biotechnol, 1999, 22(1): 1-7 |
 2016, Vol. 33
2016, Vol. 33